发布时间:2024-01-19
浏览次数:0
1. 双击打开软件包。 软件获取方法见文末。

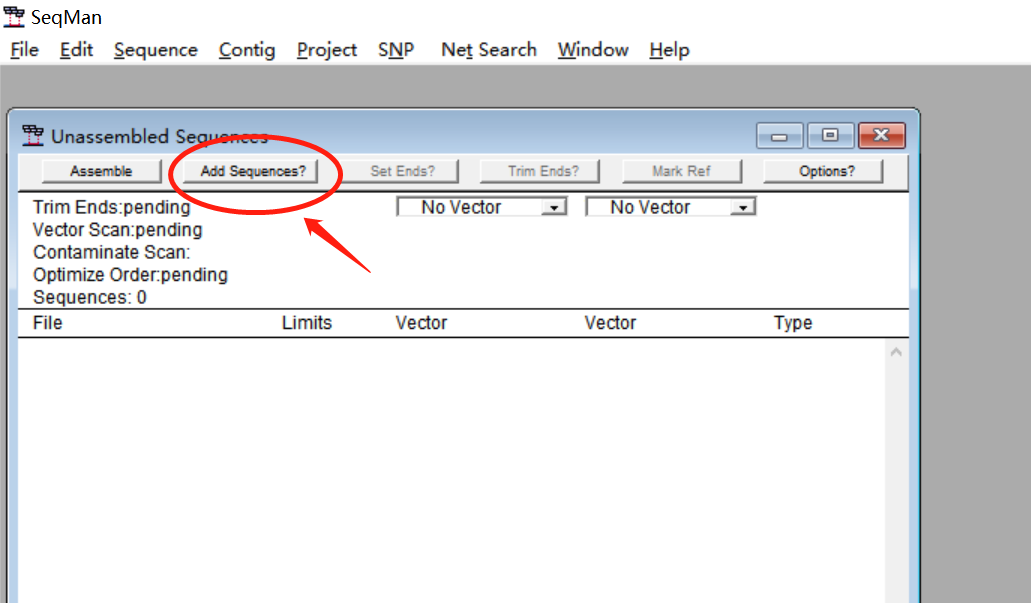
3.找到DNA序列所在的文件夹,双击需要比对的序列,然后点击“完成”
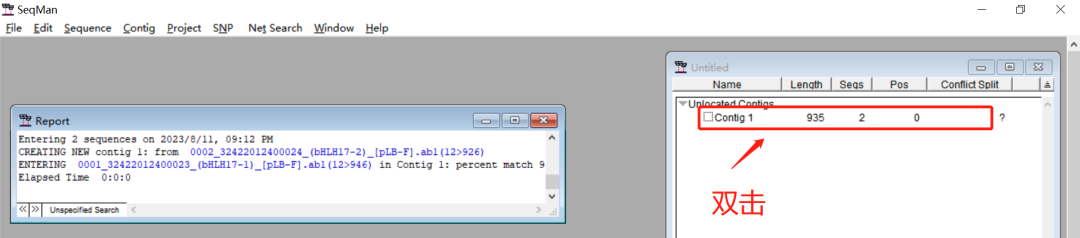
4、然后点击“”,就会出现步骤5的界面。
5. 双击出现的比较结果进行查看。 如果出现多个结果,则说明所比较的序列有较大差异。
6. 在此界面中dnastar序列比对,向左或向右拖动底部滑块可查看所有比较结果。 比较结果中出现红色的地方,说明存在差异。
7、选择测序公司提供的AB1格式文件的测序结果dnastar序列比对,也可以看到测序峰。 峰值很尖锐并且不重叠,表明信号更可靠。 序列两端出现红色片段以及重叠峰的出现并不意味着序列不正确。 可能是测序信号的问题。
选择.Seq格式测序结果文件仅查看序列而不查看测序峰。
目前的测序中,一般一个反应可以准确测量长度约为800bp的序列。
因此,如果您要测序的DNA序列小于800bp,您可以选择单向测试(正向或反向)。
如果数量大于800且小于1.6k,可以选择双向检测,这样一条DNA序列就会有正向和反向两个测序结果。
如果大于1.6k,可以选择测试一下,公司会帮你拼接。 拼接结果将是完整的DNA序列结果。 比较时,选择拼接结果中的即可。
提示:在公众号回复数字“2”即可获取.0绿色序列比对软件包。
封面图片:《1872》,莫奈
如有侵权请联系删除!
Copyright © 2023 江苏优软数字科技有限公司 All Rights Reserved.正版sublime text、Codejock、IntelliJ IDEA、sketch、Mestrenova、DNAstar服务提供商
13262879759

微信二维码