发布时间:2025-06-05
浏览次数:0
单位:黑龙江省农业科学院畜牧研究所
摘要:我国拥有丰富的野猪资源,这些野猪在生物多样性和生态系统中扮演着关键角色。深入探究野猪的系统进化,对于揭示家猪的起源与进化历程具有不可估量的价值。为此,我们在黑龙江省采集了23份野猪样本,对这些样本的线粒体细胞色素b基因以及D-loop高变区(长度为150至591碱基对)进行了详细的测序与进化研究。研究结果显示,黑龙江野猪的Cyt b基因序列遗传多样性相对较少,而不同地区的野猪亲缘关系与地理分布相吻合;在线粒体D-loop区域,目标序列中存在27个SNP位点及21个单倍型,这些序列可分为两个类群,其中类群I属于古老类群,黑龙江野猪拥有3个单倍型,涵盖了内蒙古野猪和吉林野猪的所有单倍型。据此推测,黑龙江地区可能是野猪分化的重要中心之一,这一研究为东北亚地区野猪的起源与进化提供了重要的理论支持。
野猪的细胞色素b基因序列,在mtDNA D-loop区域呈现出独特的单倍型,这一特征揭示了其系谱的进化历程。
猪是人类最早驯化的野生动物之一,经过长期的驯化过程,变成了家养动物。由于不同地域和文化的差异,全球猪种在遗传多样性上极为丰富,目前普遍认为至少有两个主要的猪种驯化中心。野猪转变为家猪是自然选择和人工选择共同作用的结果。从分子遗传学的角度来研究猪的起源和进化dnastar lasergene,可以发现人类的需求性选择对各个地区猪种群的分化程度产生了影响,导致了猪品种在表型和生产性状上出现了明显的差异。韩洪金等人通过研究酪氨酸酶基因的外显子序列,揭示了我国地方猪种是从东亚区域的野猪经过驯化而形成的。然而,目前对东亚区域野猪的进化研究还相对匮乏。鉴于此,对黑龙江野猪进行系统进化研究,对于阐述东北区域家猪的起源与进化历程具有至关重要的价值。全球野猪种类繁多,分为5个属、8个种以及23个亚种。在我国,野猪的种类相对较少,仅有一种和7个亚种。具体到地理位置,黑龙江省的野猪应当归类为中国野猪的东北亚种,学名为Sus。Alves等人通过对线粒体DNA的研究揭示,不同品种的野猪被分别驯化为欧洲和亚洲地区的家猪;Chen等人在对欧洲家猪与野猪的线粒体DNA进行研究时发现,兰德瑞斯猪与欧洲野猪的酶切图谱并无明显区别,这表明兰德瑞斯猪有可能源自欧洲野猪的驯化。黑龙江省坐落于我国东北地区,其北部和东部边界与俄罗斯接壤,而西部则与内蒙古自治区相接,南部则与吉林省相邻。在这里,黑龙江野猪与内蒙古、吉林、辽宁以及韩国的野猪之间的进化联系,一直是科研人员研究的焦点。目前在进行物种的起源、进化以及驯化研究时,线粒体DNA检测技术被普遍采用,特别是针对线粒体细胞色素b基因(b,Cyt b)以及线粒体D-loop区域的检测。伴随着高通量测序技术的进步和广泛应用,Cyt b基因和线粒体D-loop区域依然被广泛视为研究起源和进化过程中的关键标记。Cyt b基因在动植物种群中的进化速度适中,这一特性使其非常适合用于野生动物群体间的水平差异检验。研究发现,猪的Cyt b基因存在7个SNP位点,这些位点的变异对繁殖性状产生了显著的影响,据此认为Cyt b基因可以作为母猪繁殖性能的标记。Wang等人通过Cyt b基因分析山东省的地方猪品种与国外猪品种的遗传多样性和系统发育关系,结果发现不同地方猪品种之间的遗传距离相对较小,这表明它们可能源自一个共同的祖先。线粒体的D-loop区域是线粒体基因组中的调控区域,它位于tRNA-Pro和tRNA-Phe基因之间。这个区域通常包含左功能区、右功能区和中间的保守区三个部分。Larso等人的研究指出,家猪和野猪在这三个区域中遗传上的相似性较高,这从侧面印证了不同国家的家猪可能源自野猪的驯化。Hongo等人在对越南的家猪和野猪的线粒体DNA进行研究时发现,亚洲野猪和亚洲家猪共同构成了越南本土的家猪。而Kim等人则对济州猪(韩国)、中国猪、澳大利亚野猪以及欧洲猪等不同猪种进行了研究,他们基于最大似然距离,分析了这些猪种线粒体D-loop区域的序列多态性,并探讨了不同猪种之间的系统发育关系,从而揭示了不同猪种的起源和进化历程。参考已有研究成果,本研究采用Cyt b基因和线粒体D-loop区域的测序技术,结合发育学分析和单倍型组成研究,探讨黑龙江野猪的系统进化历程,这一研究方法是切实可行的。目前,对于我国东北部地区的野猪起源及进化历程的研究报道并不多见。在该地区野猪在全国野猪进化谱系中的具体位置尚未明确。鉴于此,本研究计划收集黑龙江省的野猪样本,并与NCBI提供的其他区域样本的Cyt b基因及线粒体D-loop区域数据进行整合分析dnastar lasergene,进而进行分子进化研究。这一研究的目的在于,从线粒体层面检测并解析黑龙江省野猪的系统演化过程。
1 材料与方法
1.1 试验材料
实验所用的黑龙江野猪样本共计23份,这些样本均采集自黑龙江省的小兴安岭区域,具体包括耳部组织样本,目前这些样本已被保存在液氮中,放置于黑龙江省农业科学院畜牧研究所的分子遗传实验室。
实验所需的药剂DNA、四种脱氧核苷酸(dNTPs)、rTaq DNA聚合酶以及pMD18-T载体均源自大连宝生物工程有限公司;同时,胶回收试剂盒和质粒提取试剂盒则由上海华舜生物工程有限公司提供;而其他实验用试剂均为国产或进口的高纯度分析级产品。
1.2 方法
从野猪耳组织中提取50至100毫克的样本,采用酚法进行DNA提取,并通过分光光度计及琼脂糖凝胶电泳技术对DNA质量进行测定与检验。检测结果显示,样品DNA的OD260/280比值介于1.70至1.90之间。在1%琼脂糖凝胶电泳中,需确保条带清晰、排列整齐、无拖尾现象,且点样孔附近无残留物,以满足后续实验需求。提取的DNA样本需在-20℃条件下保存,以备后续使用。
1.2.2 在设计引物时,我们参考了NCBI数据库中猪的Cyt b基因序列,并据此合成了引物(分别为:5' ATG ACC AAC ATC CGA CAC C 3';5' TCA CTT TAA TAG TTC GAT G 3'),预计通过扩增得到的片段长度为1,137个碱基对。依据NCBI数据库中猪的线粒体DNA序列信息,我们设计了相应的引物(分别为:5' CAA GCA TTC CAT TCG 3';5' TGT ACG TTA TGT CCT ATT G 3'),针对D-loop区段进行扩增,该区域长度介于150至591碱基对之间,预计扩增产物长度将达到441碱基对。
PCR反应体系包含以下成分:基因组DNA,浓度为50 ng·μL-1,取1 μL;上、下游引物,浓度为10 pmol·μL-1,各取1 μL;rTaq DNA聚合酶,浓度为5 U·μL-1,取1 μL;dNTPs,浓度为10 mmol·L-1,取2 μL;10 × PCR缓冲液,取2 μL;ddH2O,取11 μL。将这些成分混合后,总体积达到20 μL。PCR反应的参数设定如下:首先,在95℃下进行5分钟的预变性处理;接着进行30个循环,每个循环包括95℃下30秒的变性,60℃下30秒的退火,以及72℃下60秒的延伸,之后结束循环;最后,在72℃下延伸10分钟,并在16℃下继续保温30分钟。在此过程中,Cyt b基因的退火温度设定为60℃,而线粒体D-loop区的退火温度则为57℃。
对克隆测序中获得的PCR扩增产物,首先采用1%琼脂糖凝胶电泳进行目的片段的回收操作,随后将其与pMD18-T载体连接,接着转化至大肠杆菌DH5α感受态细胞中,提取得到的阳性克隆质粒,并最终将样本送至北京华大公司进行测序。
对所测序列进行拼接和人工比对,借助软件完成这一过程。随后,运用MEGA 7.0软件对整理后的序列进行比对,并生成.meg格式的文件。打开该文件时,采用DNASP 5.10软件,对比对序列中的SNP位点、单倍型数量、单倍型多样性、核苷酸多样性、平均核苷酸差异数以及保守区间等进行统计分析。
从下载的16条野猪Cyt b基因序列和457个已注释的线粒体D-loop区序列(包括D-loop数据集:: , , ,)中,结合实验室获取的黑龙江野猪序列,选取了17条Cyt b基因序列用于构建分子进化树,同时利用480条线粒体D-loop区序列进行单倍型鉴定和系统发育分析。单倍型鉴定通过Seq-Man Pro进行分离,SNP的保留标准是依据最小等位基因频率(Minor ,MAF)是否大于0.05,所有序列的比对工作均由Mega-X软件完成,进化关系的构建采用邻接法,并对每个分支的统计支持进行了500次重复分析评估。
2 结果与分析
2.1 黑龙江野猪Cyt b基因系统进化分析
利用PCR技术进行扩增和克隆测序,以黑龙江省的野猪线粒体DNA为模板,成功获取了黑龙江野猪的Cyt b基因片段。扩增结果如图1所示,其长度与预期相符。
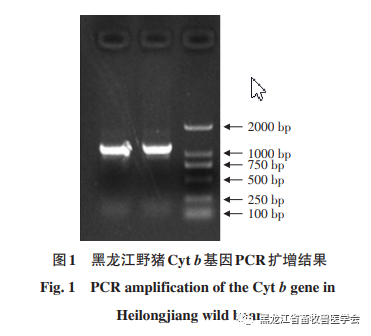
成功获取了23条黑龙江野猪的Cyt b基因序列,这些序列的Cyt b基因全长均为1140碱基对,具体数据见图2。在这23条序列中,腺嘌呤(A)、胸腺嘧啶(T)、胞嘧啶(C)和鸟嘌呤(G)的含量分别占30.2%、26.9%、29.4%和13.5%。平均而言,A+T的总含量为57.1%,C+G的总含量为42.9%,A+T的含量略超于C+G,但两者之间没有出现显著的差异。黑龙江省的野猪在23条Cyt b基因序列中发现了两个变异位置,这些位置均为简约信息位点,具体位于693碱基对和1 045碱基对的位置,且不存在单一的变异位置。通过运用Dna SP 5.1软件进行序列分析,我们得知黑龙江野猪的23条Cyt b序列中,共识别出3种不同的单倍型。这些单倍型的多样性指数(Hd)为0.605,核苷酸多样性(π)为0.00067,平均核苷酸差异数(K)为0.69203。进一步采用's D和Fu's Fs检验,结果显示黑龙江野猪的's D和Fu's Fs值均为正值,具体数值分别为0.64237和0.90198,且两者之间的差异并不显著(P>0.05)。这一结果说明,黑龙江野猪群体遵循中性进化假说,且其种群状态相对稳定。
在NCBI数据库中,我们对黑龙江野猪的Cyt b基因进行了系统进化分析,整理收集了16条野猪Cyt b基因序列数据。这些序列中,有9条来自欧洲、亚洲和非洲的野猪,另外7条则来自中国不同地区的野猪,具体信息详见表1。

通过构建不同野猪群体的Cyt b基因分子进化树,如图3所示,研究发现,欧洲与非洲野猪聚合成一个分支,而亚洲野猪则单独形成一个分支,这种亲缘关系的分布与地理分布特征相吻合。
2.2 黑龙江野猪线粒体D-loop 区系统进化分析
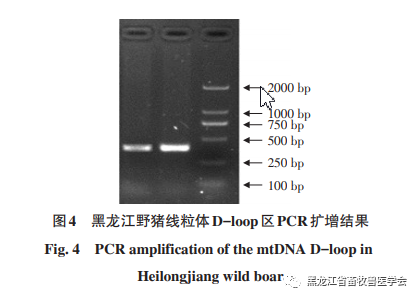
利用黑龙江野猪的线粒体DNA作为模板,我们成功进行了PCR扩增,并检测到了黑龙江野猪线粒体DNA的D-loop高变区(范围在150至591碱基对之间)的条带。具体扩增结果展示在图4中,其长度与预期相符。成功解析出23条黑龙江野猪线粒体D-loop高变区的基因序列,这些序列中腺嘌呤(A)、胸腺嘧啶(T)、胞嘧啶(C)和鸟嘌呤(G)的含量分别达到了28.7%、30.0%、25.5%和15.8%,平均的A+T含量为58.7%,而C+G的平均含量为41.3%,A+T的含量略微超过C+G,但两者之间的差异并不显著,具体序列信息可参考图5。黑龙江野猪的23条D-loop序列在280碱基对、388碱基对、453碱基对和561碱基对位置上发现了4个变异点,这些位点均为简约信息位点。通过Dna SP 5.1软件的分析,我们得知这23条序列中检测出了3种不同的单倍型,其单倍型多样性(Hd)达到了0.755,核苷酸多样性(π)为0.00377,平均核苷酸差异数(K)为1.668。进一步运用's D和Fu's Fs检验,结果显示中性检测值均为正值,分别为1.和2.,且两者之间的差异显著(P值)。

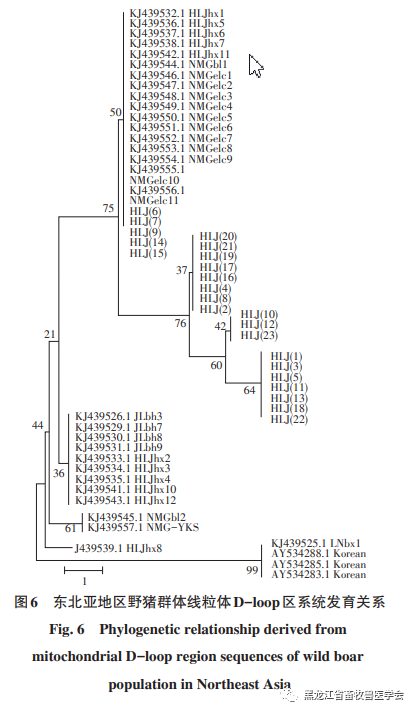
为研究东北亚野猪种群的分化情况,我们首先对NCBI数据库中的相关子库进行了整合,其中涵盖了内蒙古、吉林、辽宁以及韩国的野猪子数据集。接着,我们运用邻接法,基于D-loop区的核苷酸序列构建了分子进化树。东北亚地区野猪群体线粒体D-loop区的系统发育关系如图6所示。图6显示,东北亚野猪大致可分为三类群体,具体来看,内蒙古、黑龙江以及吉林三地的野猪归属于两个主要的群体,一个群体由黑龙江与内蒙古的野猪构成,另一个群体则包括了黑龙江、吉林和内蒙古的野猪,而第三群体则由辽宁和韩国的野猪构成。这一分布情况反映出东北亚野猪的亲缘关系与地理分布相吻合,尽管在不同地区间仍存在一定的差异。
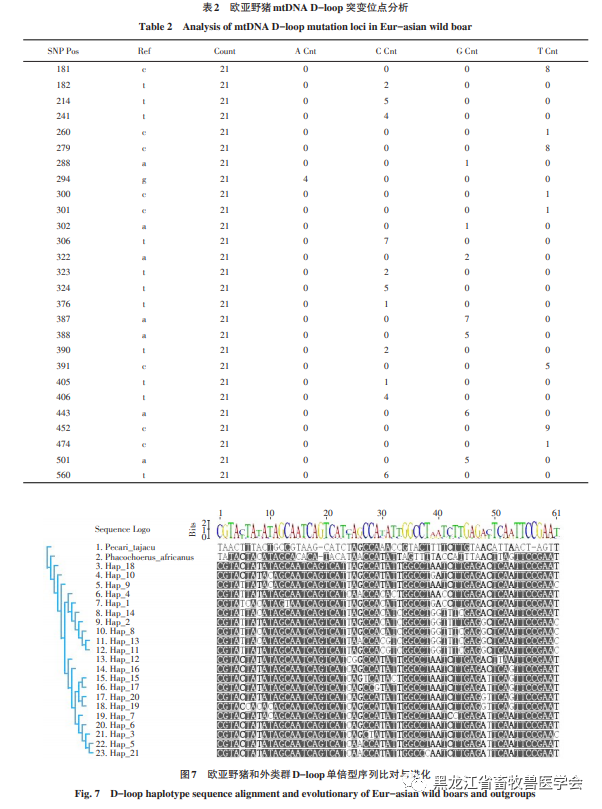
为深入探究黑龙江野猪的起源与进化,我们采用了野猪线粒体D-loop区的单倍型鉴定方法。此方法整合了NCBI数据库中已注释的西亚、欧洲、东南亚以及中国其他地区野猪的序列数据,构建了一个包含480个样本的欧亚野猪D-loop区序列集合。在猯和疣猪作为进化外类群的基础上,我们通过Pro过滤手段排除了MAF(Minor ,小等位基因频率)小于0.05的位点,最终获得了27个有效的SNPs( ,单核苷酸多态性),其中包括19个简约信息位点和8个单一变异位点。其中,最大简约信息位点为452位的C/T,根据外类群提供的信息,T位点被认为是后天突变。具体信息详见表2。这些数据共组成了21个单倍型,欧亚野猪与外类群D-loop单倍型序列的比对及进化关系可参考图7。图7显示,野猪的单倍型可以被划分为两大类,即类群I和类群II。其中,类群I涵盖了单倍型Hap 1、Hap 2、Hap 4、Hap 8、Hap 9、Hap 11、Hap 13、Hap 14以及Hap 18。而类群B则包括了单倍型Hap 3、Hap 5、Hap 6、Hap 7、Hap 10、Hap 12、Hap 15、Hap 16和Hap 17。在类群I中,除了单倍型4之外,其他单倍型均包含外类群中的代表24T和49C。黑龙江野猪群体中存在三种单倍型,它们分别是Hap 7、Hap 9和Hap 15。在这三种中,Hap 9和Hap 15属于类群I,而Hap 7则归属于类群II。
基于对欧亚野猪D-loop区序列单倍型的分析,本研究对东北亚地区包括黑龙江、内蒙古、吉林、辽宁及韩国野猪在内的56个野猪线粒体D-loop区高变序列进行了单倍型分类。研究发现,东北亚野猪种群中存在5个单倍型,并分为两个类群:类群I包含Hap 3、Hap 7、Hap 9和Hap15,而类群II则仅由Hap 1构成。通过邻接法,我们重新构建了分子进化树,具体不同地区野猪群体的单倍型分布情况可见图8。
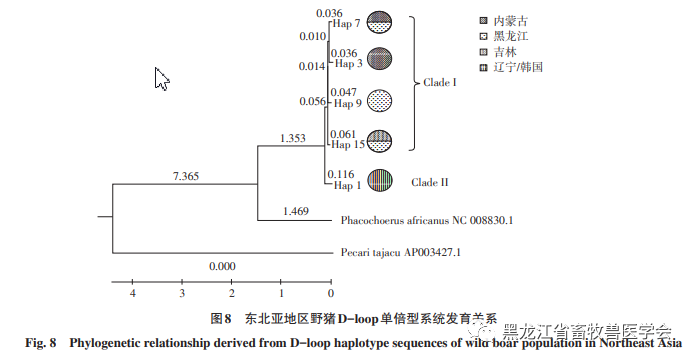
图8展示,黑龙江地区的野猪存在三种单倍型,分别是Hap 7、Hap 9以及Hap 15,并且它们携带了24C、24T和49T三个基因型;而内蒙古野猪则拥有两种单倍型,即Hap 3和Hap 7,且它们也具有24C和49T的基因型;至于辽宁和韩国的野猪,它们仅有一种单倍型,即Hap 1,并且它们的基因型24T和49C与其它类群保持一致。
3 讨 论
线粒体DNA具有母系遗传的特点,并且展现出稳定性高、背景信息明确、结构简洁、分子量较小、数据来源广泛且数量庞大等特性。这些特点使其成为研究生物母系起源的关键标记基因。此外,使用这种DNA作为试验材料的要求相对较低,且能够清晰获得研究结果。在群体遗传学、基因组学、系统与进化生物学等领域,线粒体DNA的研究应用十分广泛。然而,由于它不能反映父系遗传情况,不遵循孟德尔遗传定律,因此存在一定的局限性。以猪为例,其线粒体DNA编码了Cyt b基因等37个基因,而D-loop区域则是一个非编码区,其主要功能是调控线粒体DNA的复制和转录过程。本研究通过分析Cyt b基因以及D-loop区域,对黑龙江野猪的系统进化进行了探究,并从母系遗传的视角揭示了其演化规律。然而,这一研究同样受到了线粒体DNA局限性的影响,因此需要进一步的深入研究。
3.2 野猪的Cyt b基因系统在全球范围内广泛存在,研究人员运用线粒体DNA检测技术,对野猪与家猪的起源进行了深入研究。他们发现,不同地区的家猪在线粒体DNA上的遗传差异显著,而同一地区的家猪与野猪则拥有较多的共同遗传特征。这一发现证实了不同地区的家猪均是由当地野猪驯化而来的。在前期研究中,本课题组对民猪和大白猪的Cyt b基因进行了遗传多样性分析,结果显示,民猪的60条Cyt b基因序列中存在5个变异位置和5种单倍型,其单倍型多样性指数为0.638,核苷酸多样性指数为0.0011,平均核苷酸差异数为1.11356。中性检测进一步表明,民猪群体符合中性进化假说,种群状态相对稳定。而大白猪的Cyt b基因序列则共发现14个变异位置和4种单倍型,其单倍型多样性指数为0.581,核苷酸多样性指数为0.00599,平均核苷酸差异数为6.05419。中性检测显示,大白猪群体不符合中性进化假说,种群经历了扩张过程。本项研究在探讨黑龙江野猪Cyt b基因时,同样运用了单倍型多样性、核苷酸多样性和平均核苷酸差异数等指标来评估其遗传多样性。对比民猪和大白猪群体,我们发现黑龙江野猪的遗传多样性相对较低,其核苷酸多样性显著低于民猪和大白猪。这一现象可能与黑龙江野猪在野生状态下不同血统个体间的交流受限有关。在大白猪群体中,核苷酸多样性最高,这或许与商品化养殖为避免近交而持续引入外来血统或与其他猪种杂交改良有关。通过'sD和Fu's Fs检验,中性检测结果显示黑龙江野猪和民猪均符合中性进化假说,表明种群处于较为稳定的状态。相反,大白猪群体不符合中性进化假说,暗示其种群经历了扩张。这些研究结果与三个种群的实际情况相吻合。黑龙江野猪长期处于自然繁衍状态,民猪在市场上的竞争力不及进口商品猪种,近年来一直处于保种状态,并未经历快速扩群,因此处于相对稳定的状态。而大白猪则是目前全球范围内大规模养殖的品种。系统进化分析显示,欧洲和非洲野猪被归为一类,亚洲野猪则聚为另一类,它们之间的亲缘关系与地理分布特征相吻合。
张怡在研究野猪线粒体D-loop区系统进化时发现,野猪种群的遗传结构中,有27个单倍型是东亚野猪线粒体DNA D-loop区序列所独有的。这一发现表明,东亚野猪的遗传多样性明显超过了欧洲野猪。据此,张怡认为东亚野猪是由12个独立的分支所构成的。此外,朝鲜野猪和韩国野猪分别分布在其中的3个亚支里。而东亚野猪在不同地理区域也经历了不同的种群历史。本研究对来自黑龙江的23头野猪样本进行了线粒体D-loop高变区150至591碱基对的测序分析。分析结果显示,构建的系统进化树揭示了黑龙江野猪与吉林野猪、内蒙古野猪之间的亲缘关系较为密切,而与辽宁野猪及韩国野猪之间的距离则相对较远。这一发现与地理分布规律相吻合,并且与黑龙江野猪Cyt b基因的系统进化分析结果相一致,同时,也与其他研究者张冬杰等人对黑龙江野猪分子进化研究所得结论相符。在全球家猪的进化研究中,有一些学者倾向于接受传统的二元起源理论,该理论认为现代的家猪起源于欧洲和亚洲两个主要的母系群体,因此,野生猪种也被称作欧亚野猪。野猪依据地理和形态学的不同特征,可以被划分为四个“亚种群”:欧洲亚群涵盖了欧洲野猪、萨丁岛野猪、北非野猪、中东野猪、高加索野猪以及里海野猪;印度亚群则包括印度野猪和斯里兰卡野猪;远东亚群则由西伯利亚野猪、乌苏里野猪、日本野猪、琉球野猪、中国台湾野猪、华南野猪和高丽野猪组成;而印尼亚群则仅包含印尼野猪。截至目前,已知的野猪亚种共有20种,其中黑龙江省内的野猪,根据地理位置的划分,应当归属于远东亚群。倪攀通过研究欧亚野猪的线粒体DNA,揭示了东北野猪在欧洲家猪和野猪的谱系中占据一席之地,同时,一些欧洲家猪也出现在亚洲的谱系中,这表明了母系遗传的渗透现象。李崇奇等人对中国野猪的线粒体DNA进行了研究,指出华南地区的野猪可能通过自然迁徙或人类的迁移活动,逐渐从华南迁移至北部,最终在中国东北形成了特有的东北野猪种群。徐秋良等人在研究淮南猪和东北野猪的单倍型网络时发现,尽管东北野猪与淮南猪存在一些共同的遗传标记,但它们与国外的猪种在亲缘关系上更为接近。杨国伟等人的研究发现,东北区域的野猪或许源自欧洲的迁移。黑龙江省与俄罗斯相邻,这一地理位置使得其地域范围横跨了欧亚两大洲,因此,部分欧洲野猪得以参与到东北地区的野猪种群中。
本研究基于线粒体D-loop高变区单倍型进行筛选,发现欧亚野猪存在21种单倍型。从图6中可以观察到,外类群中具有代表性的SNP是24T和49C。据此推断,类群I应为最古老的野猪种群。而Hap9和Hap10分别对应24C和49C,它们是向类群II过渡的典型单倍型。类群II则代表了后续演化的野猪种群。这一类群可能是由第二轮进化扩散所形成,为欧亚野猪的驯化起源提供了理论支持。分析单倍型构成,东北亚野猪共有五种线粒体D-loop单倍型。图7展示了这些单倍型的分子进化关系,其中内蒙古、黑龙江和吉林的野猪归属于类群II,而辽宁和韩国的野猪则属于类群I。在黑龙江野猪的3种单倍型Hap 7、Hap 9和Hap 15中,存在24C、24T和49T变异。内蒙古野猪的2种单倍型Hap 3和Hap 7中,24C和49T变异相同。辽宁野猪和韩国野猪的唯一单倍型Hap 1,D-loop区呈现24T和49C,与外类群猯和疣猪相同,这表明辽东半岛野猪可能是最古老的野猪进化分支之一。然而,由于样本数量有限,本研究及NCBI数据库中的辽宁、吉林、黑龙江和内蒙古野猪线粒体DNA序列中,未发现过渡单倍型Hap9和Hap10,这暗示吉林、黑龙江和内蒙古野猪并非源自辽东半岛,而是可能通过其他迁徙路线形成,并在吉林、黑龙江和内蒙古等地分化。值得注意的是,黑龙江野猪的线粒体D-loop区单倍型种类最多,包括内蒙古和吉林野猪的所有单倍型,这表明黑龙江地区可能是中国东北部野猪分化的中心之一。这些研究结果为东北亚地区野猪的起源与进化提供了理论支持。
4 结 论
成功获取了黑龙江野猪的Cyt b基因序列,结果显示其遗传多样性并不高;通过分子进化树的构建,可以看出不同地区的野猪之间亲缘关系的远近与地理分布相吻合,这一结果与它们线粒体Dloop区的序列进化分析结果相一致。
黑龙江野猪线粒体D-loop区的序列分析揭示了单倍型系统的进化特征,结果显示黑龙江野猪的单倍型种类繁多,达到了三种,涵盖了内蒙古野猪和吉林野猪的所有单倍型。这一发现暗示,黑龙江地区可能是野猪分化的重要中心之一。尽管本研究综合了NCBI数据库中野猪线粒体Dloop区的众多数据,然而仍难以确保野猪样本的全面覆盖。鉴于线粒体DNA是追踪母系遗传的鉴定工具,后续研究有必要进行更大规模的样本采集以及全基因组测序,以便揭示东北亚地区野猪的真实进化轨迹。
01
作者介绍
王文涛
黑龙江省农业科学院畜牧研究所

身为中共党员、副研究员,东北林业大学农学博士、博士生,黑龙江省农科院畜牧研究所副主任,我专注于畜禽种质特性研究、饲养管理、农业废弃物处理等领域,并投身于种养结合理论研究和产业经济方向的工作。作为国家生猪产业技术体系团队的核心成员,农业部百名优秀科研创新团队的一员,以及农业部种养结合重点实验室的技术组成员,我还在国家农业废弃物处理联盟和国家畜禽废弃物综合利用技术创新联盟中担任理事。此外,我还担任华夏地方猪产业技术创新联盟的副秘书长,以及黑龙江省畜牧兽医学会和黑龙江省绿色生猪养殖协会的副理事长。在此期间,我主持和参与了40余项国家科技支撑计划、农业部产业技术体系建设项目、国际合作项目和黑龙江省科技攻关项目。同时,我出版了5本书籍,制定了6个地方标准,获得了23项专利。在科研成就方面,我荣获省科技进步一等奖1项、二等奖2项,省畜牧科技进步奖4项,畜牧丰收奖3项,并取得4项成果。
投稿单位:黑龙江省农业科学院畜牧研究所
平台主编:何鑫淼
执行主编:王文涛
如有侵权请联系删除!
Copyright © 2023 江苏优软数字科技有限公司 All Rights Reserved.正版sublime text、Codejock、IntelliJ IDEA、sketch、Mestrenova、DNAstar服务提供商
13262879759

微信二维码