发布时间:2024-01-11
浏览次数:0
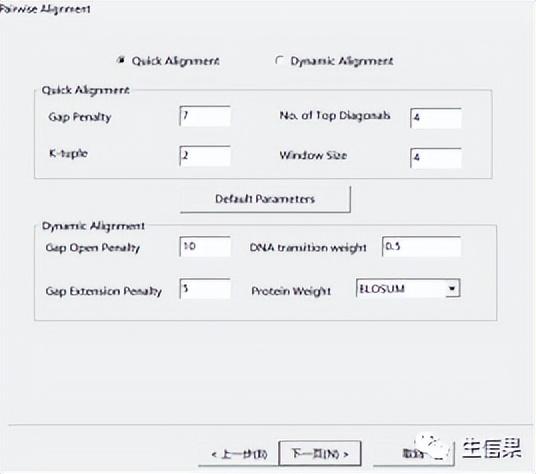
*K元组的大小一般设置在2-6之间,具体取决于序列的长度和变异性。 通常,较短的 K 元组能够更好地捕获序列中的局部相似性,但可能在比对过程中产生不匹配。 较长的 K 元组可以更好地区分序列差异,但可能会遗漏一些相似区域。
*gap和gap:这两个参数控制,即对gap出现的惩罚程度。 一般来说,gap的值应该高于gap,因为在实际的生物序列中,gap出现的概率较低。 建议初始值分别设置为10和0。
*“delay seqs%”是多序列比对中的参数。 其功能是在比对过程中控制高度分歧的序列的跳过,以减少这些序列比对结果的影响。
完成后dnastar序列比对,等待结果。 在此窗口中,您可以查看序列比对结果,包括比对分数、相似度矩阵、序列同源性等信息。
您可以使用“文件”菜单下的“另存为”选项将比较结果保存为文本文件,也可以选择结果并使用“编辑”菜单下的“复制”选项将比较结果复制到剪贴板以方便起见。 在其他软件中进行分析和处理。
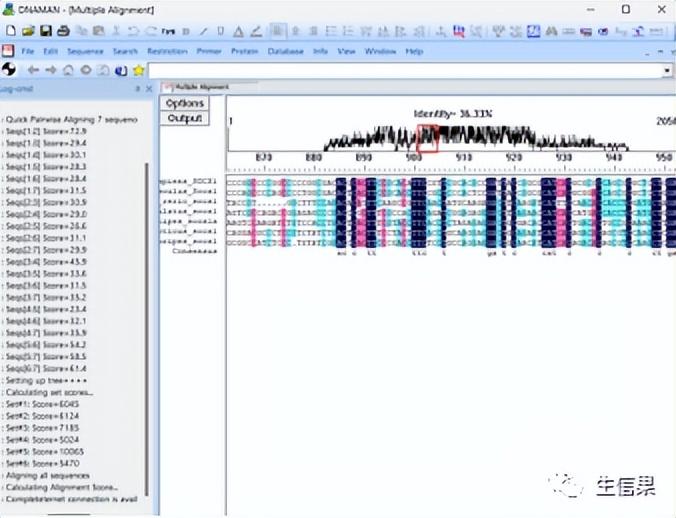
核酸比对到此结束。 您可以看到保护程度较高的区域用相同的颜色标记。 您可以通过移动上面的红框来查看不同的区域。
如果你还在为生物特征分析而烦恼,努力学习却没有领悟到要点,一定要试试这个在线平台。 无论哪种图表、小提琴图、曼哈顿图,您只需要上传数据即可!
生信人R语言学习必备
立即获取帐户
开启升级模式
(56线程、256G内存、1T个人存储)
过去的代码:
[1] 拷贝数变异下游相关性分析
【2】R可视化:——科研界的美丽图画
【3】随机森林算法用于分类预测和诊断标记筛选
【4】根据GSEA本地Java版的输出结果,将多个通道整合为一张图片
【5】基于岭回归模型和基因表达矩阵估计样本对药物反应的敏感性
【6】基于R包NMF的样本类型分析
【7】DALEX包用于探索、解释和评估模型; 分析不同特征变量对响应变量的影响
【8】基于肿瘤突变负荷TMB的KM生存分析寻找最佳
【9】基于单样本富集分析算法评估组织中免疫细胞浸润水平
【10】代码分享│什么?你也在用散点图来可视化数据之间的相关性
【11】代码分享│诊断列线图、校准曲线、决策曲线和临床影响曲线的构建
【12】代码分享│你了解基因的动态变化规律吗?
【13】代码分享│生物信息分析SCI热门图表-复杂热力图
【14】代码分享│生物信息分析SCI热门图表-火山图
【15】代码分享│生物信息学分析SCI热门图表-箱线图和小提琴图
【16】代码分享│深度学习-人工神经网络(ANN)的构建
【17】代码分享│R可视化:高分文章绘图的多类型圆图封装化绘制
【18】代码分享│R可视化:基因与功能的关系--GO功能富集网络图绘制
【19】代码分享│生物信息分析SCI热门图表—KM曲线和tROC曲线
【20】代码分享│R可视化:用R语言绘制肿瘤预后模型Cox回归分析后的森林图
【21】代码分享│生物信息分析SCI热门图表——相关热图和散点图
【22】代码分享│生物信息分析的N种R语言分析关联与可视化
【23】代码分享│获取TCGA数据很难,也不知道如何预处理,所以必须学习。
【24】代码分享│机器学习-支持向量机递归特征消除(SVM-RFE)的构建
【25】代码分享│R可视化:两个矩阵的视觉相关性分析
[26]GEO数据库多数据集差异分析集成强大工具RRA,再也不用担心去除批次效应
【27】你和生物信息学老大的距离只有2分钟,即可完成预后模型的构建和性能评估。
【28】9+SCI纯资料,模型构建中的“流量明星”,你不得不知道的LASSO
【29】一步一步教你画出漂亮优雅的套索回归模型图,为你的SCI增光添彩。
【30】R可视化:比较组间GO富集图的包
【31】代码分享| R 可视化:复杂热图绘制技术:向热图添加直方图
【32】代码共享——基于基因突变信息分析肿瘤突变负荷
【33】代码分享│无法丰富想要的途径? 不要放弃,尝试 GSEA
【34】代码分享│你还在用PCA进行降维聚类吗?最强降维模型tSNE--你值得拥有
【35】代码分享│GSVA:原来功能通路也可以用于差异分析!
【36】代码分享│:单细胞准时序分析还有你不知道的事
【37】基于基因功能注释信息挖掘关键基因
【38】基于癌症分类预测的标记特征提取的SVM-RFE分析代码
[39] 基于无监督聚类算法的基于表型数据的研究人群的层次聚类分析
【40】基于鲁棒排序整合算法整合并可视化多个数据集
【41】根据基因表达谱估计样本免疫基质评分和肿瘤纯度
【42】LASSO算法回归模型图自动绘制
【43】DCA分析对临床诊断和临床决策的影响
【44】一致性指数软件,用于根据样本预后生存信息和临床因素评估不同模型
[45] 用于探索、解释和评估模型的 DALEX 残差分析软件
【46】基于细菌群落功能丰度结果的差异功能分析与可视化
【47】根据遗传差异分析结果绘制其在染色体上的分布
【48】利用逐步回归方法筛选特征基因并构建Cox风险模型分析
【49】肿瘤免疫亚型的分类依据
【50】不同物种间同源基因名称转换分析
【51】筛选预后标志基因并构建基于逐步多因素cox回归的风险评分模型
【52】根据表达信息挖掘与目的基因密切相关的基因
【53】基因组学基因名称修正分析
【54】基于算法构建关联网络
【55】基于线性建模方法的代谢组和转录组数据整合分析
[56]基于Lasso回归模型方法筛选特征基因
【57】基于线性建模方法的代谢组和转录组数据整合分析
[58]基于参数经验贝叶斯算法和支持向量机(SVM)的疾病亚型特征基因筛选
【59】基于LDA(线性判别分析)算法的微生物筛选
【60】基于R包xCell的64种免疫细胞相对含量计算及下游可视化
【61】基于甲基化数据的肿瘤纯度评估和下游可视化
【62】基于不同表型下差异共表达关系对的包识别
【63】基于逆累积分布函数的显着偏差路径识别
【64】根据差异基因对通路的影响挖掘关键通路
【65】基于高通量数据的样本相似度分析
如果需要以上代码请私信小果!

《生物信息学果子》dnastar序列比对,生物信息学入门、R语言、生物信息学图表解读与绘制、软件操作、代码复现、生物信息学硬核知识与技能、服务器、生物信息学教程以及基于R的分析与可视化等原创内容,见证小白和老板一起成长。
如有侵权请联系删除!
Copyright © 2023 江苏优软数字科技有限公司 All Rights Reserved.正版sublime text、Codejock、IntelliJ IDEA、sketch、Mestrenova、DNAstar服务提供商
13262879759

微信二维码