发布时间:2023-10-31
浏览次数:0
提示:
1、基因测序可明确诊断,消除污染;
3.生活,生活就是ATCG。
在之前关于病原体检测技术的推文中,我们介绍了PCR检测是确定病原体的有力工具。 但PCR中的阳性条带可能是非特异性扩增dnastar序列比对,需要测序验证; 基因测序可以确诊并消除污染; 但更重要的是,测序和序列比较可用于确定菌株种类和关系。 先不说目前流行的NGS(next,深度测序)方法进行诊断。 我们只会介绍常规PCR后的直接测序或克隆测序。
测序方法主要采用化学裂解法(MAxam-)——基本不再使用。
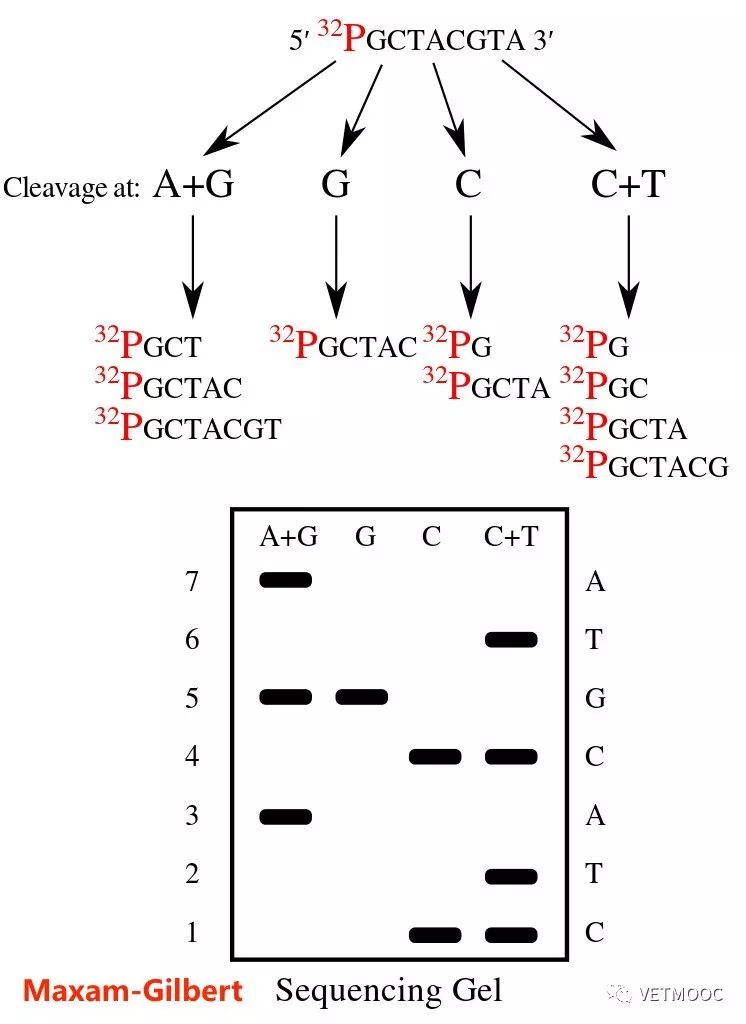
以及DNA链末端合成终止方法(方法)。 它利用了双脱氧核糖核酸(ddA、ddT、ddC 和 ddG)在添加到 DNA 链后终止延伸的特性。 基于核苷酸起始于固定点并随机终止于特定碱基的事实,并在每个碱基后面进行荧光标记,生成一系列以A、T、C、G结尾的四组不同长度的核苷酸,然后在尿素变性的 PAGE 凝胶上进行电泳检测以获得可见的 DNA 碱基。 一种碱基序列的方法。

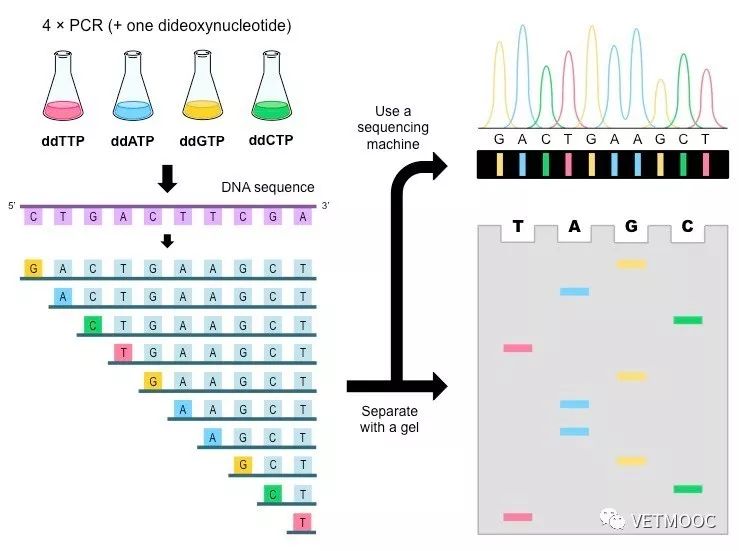
对该方法进行优化后,使用荧光代替放射性核素标记是自动化DNA序列分析的基础。 自动DNA测序仪可以用不同的荧光分子标记四种双脱氧核苷酸,然后进行测序反应。 反应产物通过电泳(平板电泳或毛细管电泳)分离后,用四束激光激发不同大小的DNA片段上的荧光分子。 它发出四种不同波长的荧光,检测器收集荧光信号并据此确定DNA碱基的序列。
说了这么多,其实现在实验室里的少男少女们基本上都是打电话让测序公司的小伙子来冰箱里取样本(一般是PCR产物)。 该公司将负责测序的凝胶鉴定和回收。 第二天,该序列就会发送到电子邮件中。

关于测序结果返回后如何分析病毒的亲缘关系,我们实验室检测组组长张新辉写了详细的操作,并做了一些编辑和修改,分享如下:
可用的序列分析软件有多种类型,常用的是软件和软件包。 用于查看测序峰、判断测序质量、截取有效序列片段; 分为 、 、 、 等多个软件,常用在检测结果的序列比对过程中。

下面用一个carry-in的场景来描述序列分析和比对的过程:
通常,将样本发送给测序公司几天后,您就会收到如下所示的测序结果:
下载电子邮件中的附件
解压后,您会发现送检的样本有两种格式的文件。
AB1后缀的文件是测序结果的峰图,可以用软件打开。 通过峰图可以知道测序结果的准确性以及准确的面积。
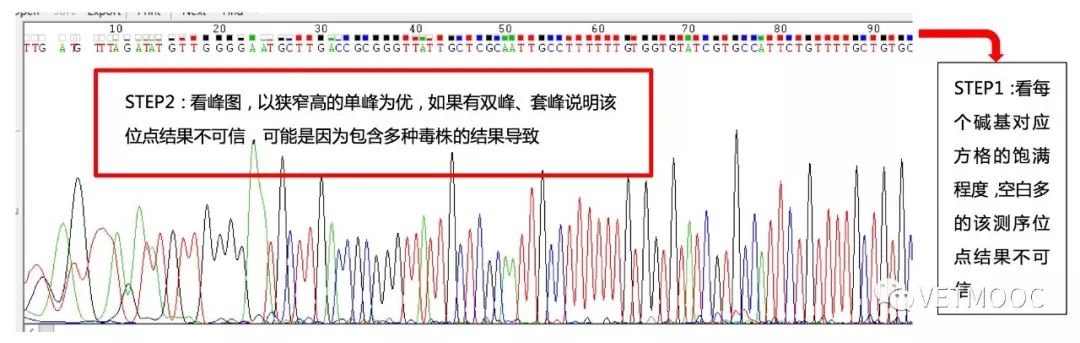
一般情况下,方法1和方法2结合使用。 通常情况下,测序结果的头峰和尾峰比较杂乱,序列不准确。 在序列比对过程中,我们一般要先将测序结果的头尾序列片段剪掉再进行分析。 做PCR测序时,需要流出足够的侧翼序列,使得PCR片段设计得比实际需要测序的片段大,这样才能保证获得足够完整的基因序列。 如果将PCR产物克隆到载体中,利用载体上的通用引物进行测序,就可以保证之前不准确的区域都是载体序列,而且插入序列时峰型会很整齐,不会出现重复的情况。影响测序结果。
我们通常会打开SEQ文件,以便看到测序结果并进行进一步的编辑操作。
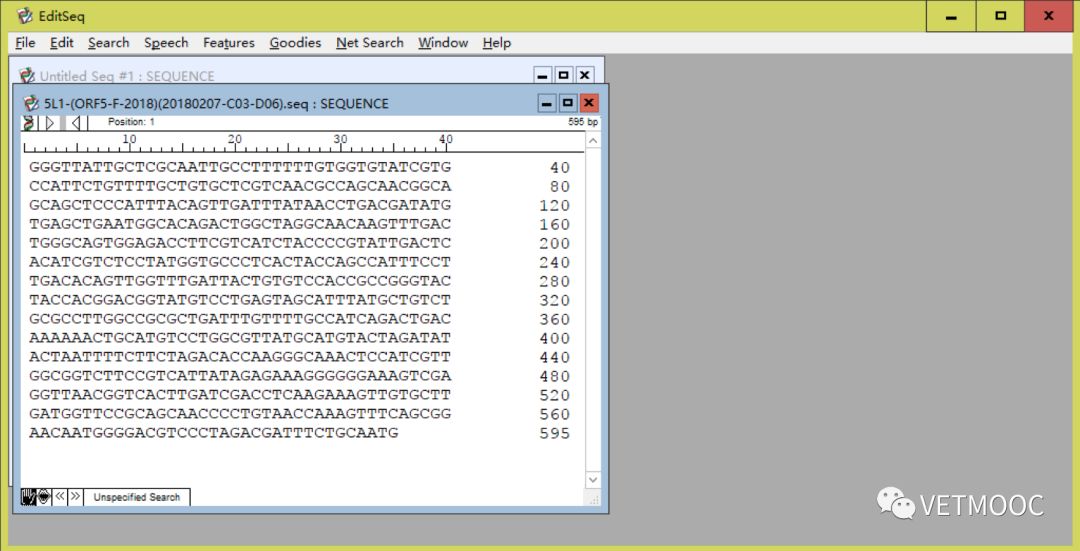
我们根据峰值图的结果删除SEQ文件中首尾不稳定的片段,并保存编辑后的文件(截断头部和尾部,根据峰值图的整洁程度选择截断的序列)

用名称保存文件。
保存到您可以轻松找到的位置。
测序结果处理后,利用软件与国内现有的代表性菌株进行核酸相似度比对。
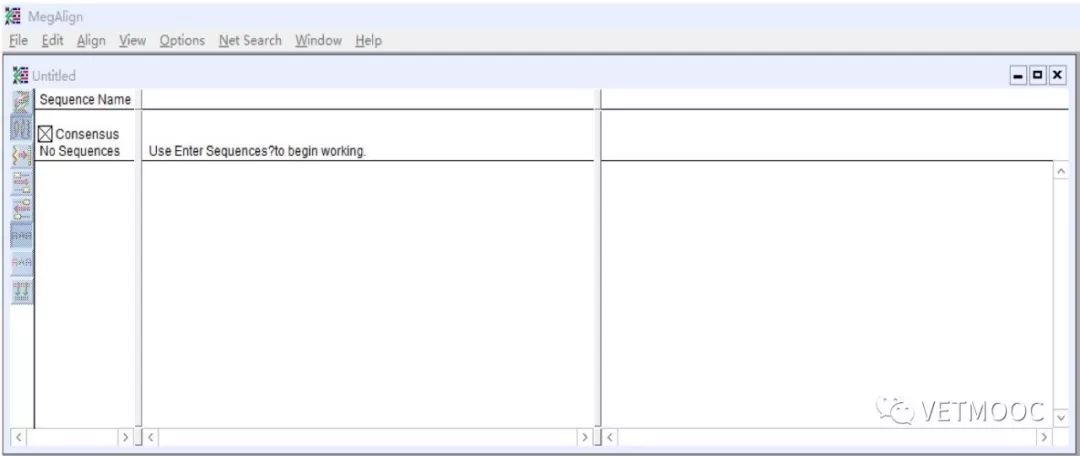
将代表菌株和测序结果的SEQ文件拖入软件中,上下拖动可调整序列顺序。
选择“对齐”>“按W”(W比较时间更长,更准确); 如果你想看氨基酸序列的同源性,可以先将核苷酸序列翻译成蛋白质,然后进行比较,前提是核苷酸序列按3个碱基排列正确,并从第一个开始位,否则会发生移码,导致翻译失败或过早终止。

软件开始自动进行一一比较。 序列的长度、数量和计算机配置将决定完成它需要多长时间。
比对完成后,选择“查看”>“”即可显示序列之间的同源性。

以PRRSV的ORF5测序为例,对比结果如下。

比较后,制作一个表格来统计相似度。
通过PRRSV测序结果与参考毒株序列的对比,可以大致看出毒株的来源。 一般情况下,PRRSV测序时选择的参考株应包括北美原型株VR-2332、国内经典株CH1-a、HP-PRRSV株JXA1(HuN4和HuN4均可)、HP-PRRSV衍生株等。 MLV JXA1-R(P80)(或类似)菌株以及新流行的QYYZ和GM2。 如果欧洲毒株LV与美国毒株的亲和力很低,则进行比较。
--谢谢大家dnastar序列比对,奖励和召唤是前进的动力--
如有侵权请联系删除!
Copyright © 2023 江苏优软数字科技有限公司 All Rights Reserved.正版sublime text、Codejock、IntelliJ IDEA、sketch、Mestrenova、DNAstar服务提供商
13262879759

微信二维码