发布时间:2023-11-12
浏览次数:0
基于NGS检测方法
融合检测软件总结
基于NGS的基因融合检测方法
基于NGS方法的基因融合鉴定技术主要有两种,1.基于DNA水平的融合检测dnastar序列比对,如全基因组测序数据(whole-测序数据(WGS)),2.基于DNA水平的融合检测在RNA水平上,如转录组测序数据(RNA-seq),3种或两种技术的结合效果更好。
基于DNA水平的融合检测
DNA测序还可以检测融合基因。 融合基因是结构变异(SV)的一种。 利用DNA测序分析融合基因的策略主要包括:
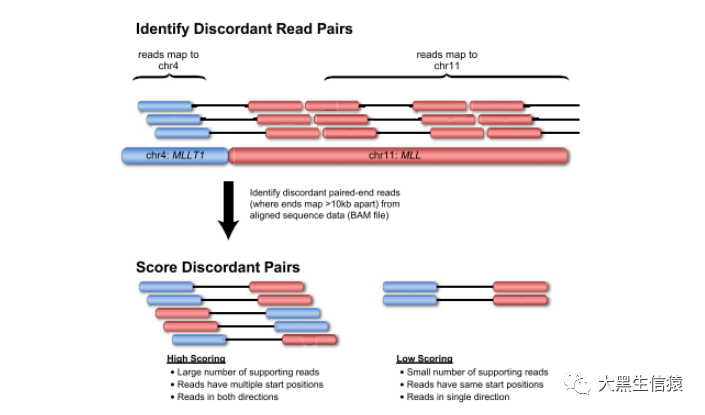
① 双端序列配对法(pair-end)
利用DNA测序分析融合基因的策略主要包括:双端序列配对法(pair-end)。 该方法的总体思路是首先将双端序列与基因组进行比对,然后评估双端序列的距离和方向是否一致。 数据库创建信息一致。
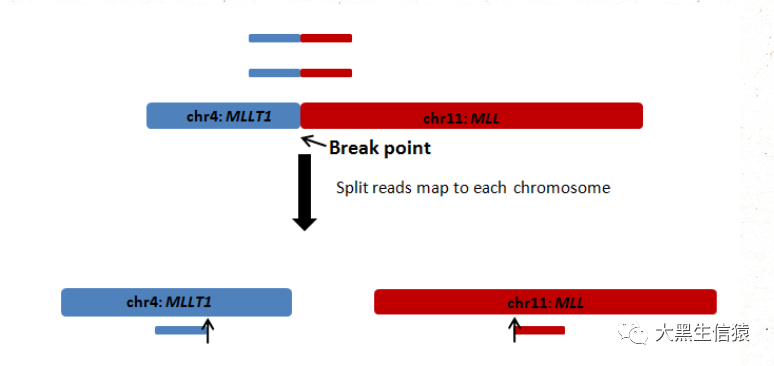
②分读法
第一步,获得与基因组比对的唯一单端序列,将序列中无法比对的部分剪掉,然后重新比对,最终得到比对位置和比对方向。
海莉·J 和埃里克. 来自下一个数据的 DNA:a of . 热内特. 2013, 206(12): 432-440。
基于全基因组测序鉴定出的基因融合的优势基本上可以确定是由基因组水平上的某种突变引起的。 其缺点是无法准确判断融合后产生的新基因能否表达或表达水平。
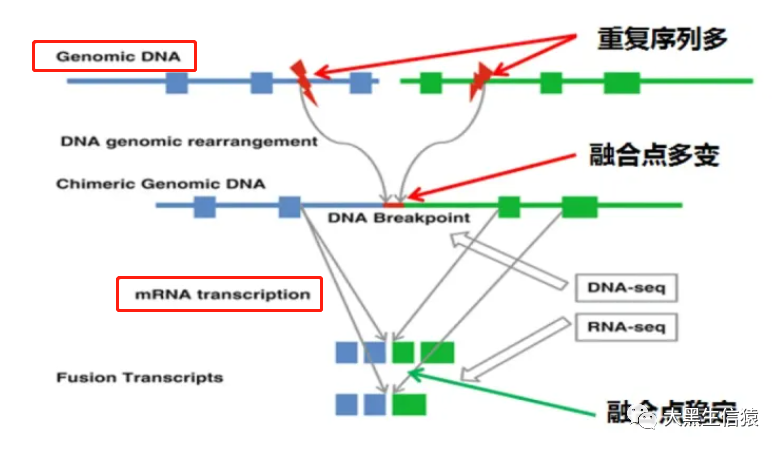
在RNA水平上,融合基因表现为前后两个基因外显子之间的连接,融合点相对固定。 在DNA水平上,融合断点通常发生在较长的内含子区域,这不可避免地具有以下局限性和挑战:
1)融合基因检测必须覆盖很长的包含大量重复序列的内含子区域,才能准确找到融合断点;
2)GC含量高不利于探针有效捕获目标区域的片段;
3)不同基因的内含子含有非常相似的重复序列。 该特性不利于准确的序列比对,影响检测精度;
4)复杂的转录或转录后剪接过程可能会影响基因融合。 因此dnastar序列比对,不难理解,在RNA水平上检测融合基因是相对高效和准确的。
基于RNA水平的融合检测
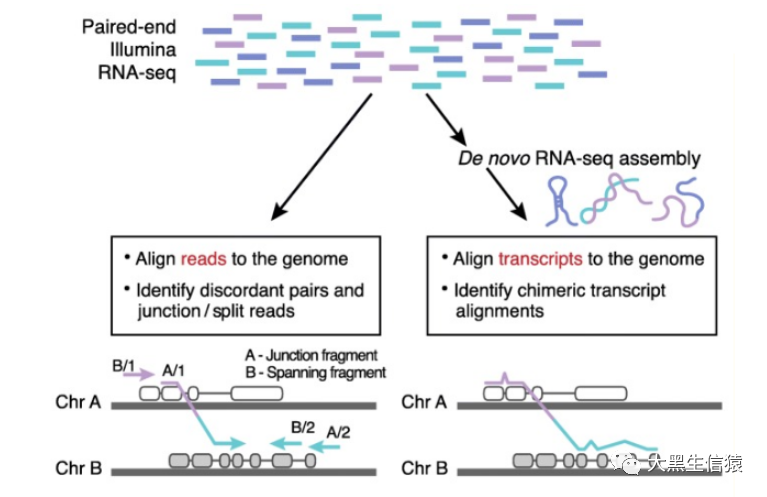
通过RNA测序检测融合基因主要有两种分析策略:
① 基于序列比较的方法,寻找不一致的序列(Pair Reads)和覆盖断点的序列(/Split Reads)来识别融合事件。
②基于剪接比对方法,首先组装新的转录本,然后与基因组进行比较,以鉴定与染色体重排一致的融合转录本[3]。
Brian J,Dobin,Bo Li,等。 基于读取和从头开始。 生物学,2019,20(1):213。
基于转录组测序数据鉴定的基因融合的优点是它们是可表达的基因融合。 缺点是不能完全确定是基因组变异引起的还是两个不同基因转录后发生的RNA融合引起的。
基因融合分析软件整理
对于软件应用,详细请参阅软件使用说明。
基于DNA检测融合软件
软件:
Git :-/: 基因和
文献:: DNA 数据中的基因 - (nih.gov)
软件:LUMPY
Git: - arq5x/lumpy-sv:lumpy:a for
文献:LUMPY:a for | | 全文 (.com)
软件:黛丽
Git : - /delly: : by-end 和 split-read
文献:DELLY:by-end 和 split-read | | (oup.com)
软件:
git:
文献:: a for the at | | (oup.com)
基于RNA检测融合软件
基因:RNA-Seq 数据 (.org)
1.:
2.:
3.:
4.:
5.:
6.:
7.:
8.机房空调:
9.:
10.:
11.:
12.:
13.:
14.:+html
15.:
16.:
17.:
18.:
19.:
20. 国内流离失所者-:
21. i保险丝:
22。 :
23。 :
24. 雅法:
25.:
26.:
27.莫乔
28.n保险丝:
29.:
30.普拉达:
31.:
32.-FTD:
33.:
34.:
35.明星:
36.星-:
37.-:
38.特鲁普:
39.:
基于全基因组和转录组测序数据的检测融合
软件:
Git:/Wiki/Home (.net)
文献::使用整体和数据的基因 - (nih.gov)
基于单细胞
软件:
单细胞基因融合检测算法
git:-/
文献:-细胞基因|
备注:子刊 | 北京大学奚瑞斌课题组研发首个单细胞基因融合检测算法——SEQ.CN (.cn)
如有侵权请联系删除!
Copyright © 2023 江苏优软数字科技有限公司 All Rights Reserved.正版sublime text、Codejock、IntelliJ IDEA、sketch、Mestrenova、DNAstar服务提供商
13262879759

微信二维码